- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Ácido aspártico - Aspartic acid
El ácido aspártico o su forma ionizada, el aspartato (símbolos Asp y D) es uno de los veinte aminoácidos que pueden componer las proteínas. En el ARN se encuentra codificado por los codones GAU o GAC.
Ácido glutámico - Glutamic acid
El ácido glutámico, o en su forma ionizada, el glutamato (abreviado Glu o E) es uno de los 20 aminoácidos que forman parte de las proteínas. Es crítico para la función celular y no es nutriente esencial porque el ser humano puede sintetizarlo a partir de otros compuestos.
Acrocéntrico - Acrocentric
Tipo de cromosoma con el centrómero cercano a un extremo. Los cromosomas acrocéntricos en la especie humana son el 13, 14, 15, 21 y 22. Contienen brazos cortos satélites que albergan genes codificantes para el ARN ribosómico.
Adaptador (relativo a secuenciación masiva) - Adapter
Cebador que se une a los extremos 5´y 3´ de cada fragmento de ADN en una librería de secuenciación. Actúa en la amplificación y secuenciación del ácido nucleico adyacente.
Adenina - Adenine
ADN (Ácido desoxirribonucleico) - DNA ( Deoxyribonucleic acid)
Es la molécula portadora la información genética, que posibilita su transmisión de una generación a la siguiente.
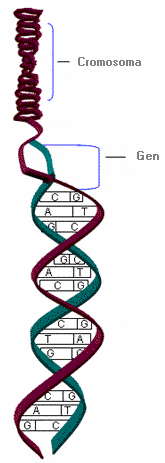
El ADN está constituido por dos cadenas de nucleótidos complementarias unidas entre sí por puentes de hidrógeno, enrollada la una sobre la otra formando una doble hélice. El elemento esencial del ADN es el nucleótido formado por un azúcar, la desoxirribosa, una base nitrogenada y un grupo fosfato. Las bases nitrogenadas son purinas: Adenina (A) y Guanina (G), y pirimidinas: Timina (T) y Citosina (C). Estas bases se unen al carbono 1 de la desoxirribosa para formar los nucleósidos, que a la vez se unirán con los grupos fosfato para formar los nucleótidos. Las bases nitrogenadas se unen de manera específica, guanina con citosina y adenina con timina, mediante puentes de hidrógeno.
El orden o secuencia de nucleótidos es la estructura primaria del ADN. La información genética reside en la secuencia de bases. Los genes son segmentos del ADN que codifican para proteínas. El orden de las bases en el gen determina la secuencia de aminoácidos que compondrán la proteína.
En las células eucariotas, el ADN se encuentra en el núcleo celular formando los cromosomas.
ADNc - cADN
ADN complementario, generado a partir del molde de ARN, mediante la retrotranscripción del ARNm maduro.
Afectado - Affected
Individuo que manifiesta síntomas de una enfermedad o patología determinada.
Alanina - Alanine
Alanina (Ala o A) es uno de los aminoácidos que forman las proteínas de los seres vivos, el más pequeño después de la glicina y se clasifica como hidrófobico. Es un aminoácido no esencial para el ser humano pero es de gran importancia y es codificada por los codones GCU, GCC, GCA y GCG.
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
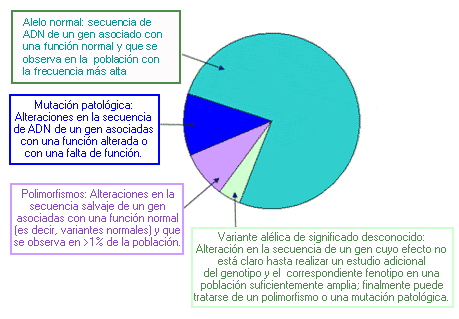
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
Alelo de penetrancia completa - Full penetrance allele
Alelo que tiene una repercusión fenotípica en todos los individuos que son portadores de ese alelo.
Alelo de penetrancia incompleta (o reducida) - Incomplete (or reduced) penetrance allele
Alelo que no tiene una repercusión fenotípica en todos los individuos que son portadores, tan solo en una porción de ellos.
Alelo normal - Wild type alelle
El asociado al fenotipo normal, el más común en la población. Opuesto a alelo mutante. También llamado “alelo salvaje” o alelo silvestre”.
Alelo nulo - Null allele
Alelo que da lugar bien a la ausencia de producto génico, o a un producto sin actividad. Una deleción del gen es necesariamente un alelo nulo.
Alelo premutado - Mutable normal allele (sinónimos: alelo intermedio, premutación)
En trastornos causados por expansión de repeticiones de nucleótidos, alelo que no está asociado con síntomas clínicos, pero puede expandirse hasta convertirse en un alelo de gran tamaño con repercusión clínica. También denominado “premutación” o “alelo intermedio”.
En trastornos causados por expansión de repeticiones de nucleótidos, el alelo permutado no está asociado con síntomas clínicos. Su tamaño es superior al del alelo normal y puede expandirse cuando es transmitido a los descendientes, transformándose en una alelo mutado.
De www.acmg.net/Pages/ACMG_Activities/stds-2002/HD.htm (con acceso 17-12-03)
Alfa-fetoproteína (AFP) - Alpha-fetoprotein
Glicoproteína fetal que se excreta al líquido amniótico y alcanza niveles anormalmente altos en el mismo cuando el feto tiene ciertas anormalidades, especialmente defectos en la abertura de tubo neural.
Alineamiento de secuencias - Sequence alingment
Alteración de la secuencia - Sequence alteration: (sinónimo: mutación)
Cualquier cambio producido en el adn[o mas concretamente en un gen con respecto a su estado natural. Si no tiene repercusión clínica se denomina polimorfismo; cuando se trata de un cambio que tiene o puede tener repercusión clínica se suele denominar mutación. Cuando no se conoce si el cambio tiene o no repercusión clínica se habla de cambio o variante de significado desconocido.
Alu, Secuencias repetidas Alu - Alu, Alu element
Secuencias de ADN repetidas distribuidas a lo largo del genoma. Son las secuencias repetidas más frecuentes, representando el 5% del genoma humano. Su nombre se debe a que son reconocidas y cortadas por la enzima de restricción Alu.
Aminoácido - Amino acid
Un aminoácido es una molécula orgánica con un grupo amino (-NH2) y un grupo carboxilo (-COOH). Los aminoácidos son compuestos orgánicos que se combinan para formar proteínas. Los aminoácidos se clasifican en tres grupos: esenciales, no esenciales y condicionales.
Los 20 aminoácidos son:
Glicina (Gly, G)
Alanina (Ala, A)
Valina (Val, V)
Leucina (Leu, L)
Isoleucina (Ile, I)
Prolina (Pro, P)
Serina (Ser, S)
Treonina (Thr, T)
Cisteína (Cys, C)
Metionina (Met, M)
Asparragina (Asn, N)
Glutamina (Glu, Q)
Fenilalanina (Phe, F)
Tirosina (Tyr, Y)
Triptófano (Trp, W)
Lisina (Lys, K)
Arginina (Arg, R)
Histidina (His, H)
Aspartato (Asp, D)
Glutamato (Glu, E)
Aminoácidos, Secuencia de, - Amino acid sequence
Amniocentesis - Amniocentesis
Técnica de diagnóstico prenatal que utiliza las células del líquido amniótico para determinar el cariotipo del feto y realizar estudios bioquímicos.
Amniocito - Amniocyte
Células fetales obtenidas por amniocentesis.
Amplicón - Amplicon
Fragmento de ADN amplificado por la reacción en cadena de la polimerasa (PCR) o cualquier otro proceso que dé lugar a la producción de diferentes copias de ese fragmento.
Amplificación - Amplification
Cualquier proceso que causa la replicación de una secuencia concreta de ADN en cantidad desproporcionadamente elevada.
Amplificación puente (relativo a secuenciación masiva) - Bridge amplification
Generación de copias in situ de una molécula específica de ADN en un soporte sólido cubierto de cebadores.
Anafase - Anaphase
Es la tercera fase de la mitosis y meiosis en la que los cromosomas duplicados son separados. Las cromátidas son entonces desplazadas a polos opuestos de la célula en división por el huso mitótico o meiótico, para que cada célula hija herede una copia de cada cromosoma.
Análisis basados en ADN - DNA-based analysis
Pruebas que implican el estudio de ADN mediante un análisis de ligamiento, secuenciación o cualquiera de los diferentes métodos utilizados para la detección de mutaciones.
Análisis de dosis - Dosage analysis
Método para determinar la cantidad de diversos componentes, incluyendo ADN, ARN y proteínas, por comparación con un estándar conocido; se puede utilizar para determinar el número de copias de una secuencia de ADN (es decir, para investigar posibles mutaciones por duplicación y deleción), mediante la comparación visual de la intensidad de las bandas o la cuantificación numérica con densitometría. Si un gen presenta copias adicionales, la intensidad es de más del 100% en el gel o película; mientras que si se ha perdido una copia del gen, la intensidad es de aproximadamente un 50%.
Análisis de fragmentos de restricción de longitud polimórfica - Restriction fragment length polymorphism analysis (sinónimo: análisis RFLP, estudios RFLP)
Estudio de los fragmentos de ADN de tamaño previsible originados por la digestión (corte) de una cadena de ADN por una enzima de restricción específica. Las alteraciones (mutaciones o polimorfismos) producidas en la secuencia de ADN que destruyen o crean nuevos sitios de corte en el ADN, modifican el tamaño (y el número) de los fragmentos de ADN resultantes de la digestión por una enzima de restricción específica.

Análisis de ligamiento - Linkage analysis (sinónimo: análisis indirecto del ADN)
Estudio de los polimorfismos de las secuencias de ADN (variantes normales) que están próximos a/o dentro de un gen de interés (ligamiento) para identificar, en una misma familia, la segregación de una mutación patológica en un gen determinado.
EXPLICACIÓN:
Las tres fases del análisis de ligamiento son las siguientes:
1. Establecer los haplotipos de cada individuo: Se analizan los múltiples marcadores de ADN dispuestos a cada lado (marcadores flanqueantes) o dentro (marcadores intragénicos) de la región del gen de interés, con objeto de determinar el conjunto de marcadores (haplotipos) de cada miembro de la familia.
2. Establecer la fase, comparando los haplotipos entre los miembros de la familia: Mediante la comparación de los haplotipos de los miembros de la familia con un estado genético conocido (es decir, afectados, no afectados), se puede identificar el haplotipo asociado con el alelo patológico.
3. Determinar el estado genético: Una vez establecido el haplotipo asociado con la enfermedad, es posible determinar el estado genético de los miembros de una familia de riesgo.
Ejemplo: Análisis de ligamiento para el diagnóstico prenatal de una enfermedad ligada al cromosoma X.
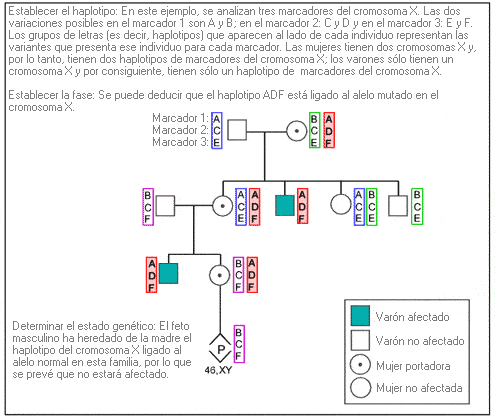
Algunas implicaciones clínicas
El análisis de ligamiento se utiliza generalmente cuando el análisis de ADN directo no es viable, porque se desconoce el gen de interés o no se puede detectar una mutación en ese gen en una familia específica.
En la mayoría de los casos, el haplotipo en sí carece de significación; sólo tiene sentido en el contexto de un estudio familiar. Sólo en el caso raro de que el desequilibrio del ligamiento sea un haplotipo específico, habrá una probabilidad muy elevada de que esté asociado con una mutación patológica específica.
La exactitud del análisis de ligamiento depende de:
La exactitud del diagnóstico clínico en un miembro o miembros de la familia que estén afectados.
La distancia entre la mutación patológica y los marcadores. El análisis de ligamiento puede proporcionar resultados falso positivo o falso negativo si, durante la formación de los gametos (meiosis), se produce recombinación que implique a los marcadores y suponga un intercambio entre el material genético de los cromosomas heredados de la madre o del padre. El riesgo de recombinación es proporcional a la distancia entre la mutación patológica y los marcadores. El riesgo de recombinación es más bajo si se utilizan marcadores intragénicos.
La informatividad de los marcadores genéticos en la familia del paciente. Si la secuencia de ADN de una variante determinada difiere entre los cromosomas heredados de la madre y del padre, este marcador es informativo. Por el contrario, si la secuencia de ADN de una variante determinada no difiere entre los dos cromosomas, el marcador no es informativo.
Análisis de matrices de ADN - DNA microarray analysis
Análisis de metilación - Methylation analysis
Métodos utilizados para evaluar el estado de metilación de un gen (unión de los grupos metilo a las citosinas del ADN). Los genes metilados no se expresan. La metilación desempeña un papel importante en la inactivación del cromosoma X y en la impronta genética.
1. Análisis con enzimas de restricción sensibles a metilación: El ADN es digerido (cortado) por una enzima de restricción que sólo puede digerir ADN no metilado. Se realizan análisis adicionales (por ejemplo, Southern blot y/o PCR) para determinar si el ADN se ha cortado. La presencia de ADN no cortado indica que el ADN está metilado, mientras que la presencia de ADN cortado indica que éste no está metilado.
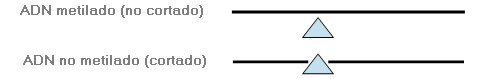
2. Análisis de secuencia tratada con bisulfito: El ADN es tratado para sustituir todos los nucleótidos citosina no metilados por nucleótidos timina. Se secuencia el ADN tratado. El ADN metilado conserva su secuencia original (normal), mientras que el ADN no metilado presenta una secuencia anormal en la que todos los nucleótidos citosina (C) se han sustituido por nucleótidos timina (T).

Análisis de microsatélites - Microsatellite analysis
Uso de secuencias repetitivas muy variables, que se encuentran en las regiones microsatélites adyacentes a los genes o intragénicas o en otras áreas de interés, como marcadores para el análisis de ligamiento, huellas moleculares de adn u otras aplicaciones diagnósticas.
Análisis de proteínas - Protein analysis
Uno de los diferentes métodos analíticos que revela la estructura o la función de un producto proteico determinado.
Análisis de replicación - Replication analysis (sinónimos: estudio de inactivación del cromosoma X)
Técnica citogenética basada en procedimientos de bandeo específicos (bandeo por replicación) para identificar el cromosoma X de replicación tardía (inactivo) en las células. En la actualidad, los estudios moleculares se utilizan con más frecuencia que el análisis de replicación para el estudio de inactivación del cromosoma X, puesto que son menos complicados, costosos y subjetivos.
Análisis de secuencias - Sequence analysis (sinónimo: secuenciación génica, secuenciación)
Proceso mediante el cual se determina la secuencia de nucleótidos en un segmento de ADN.
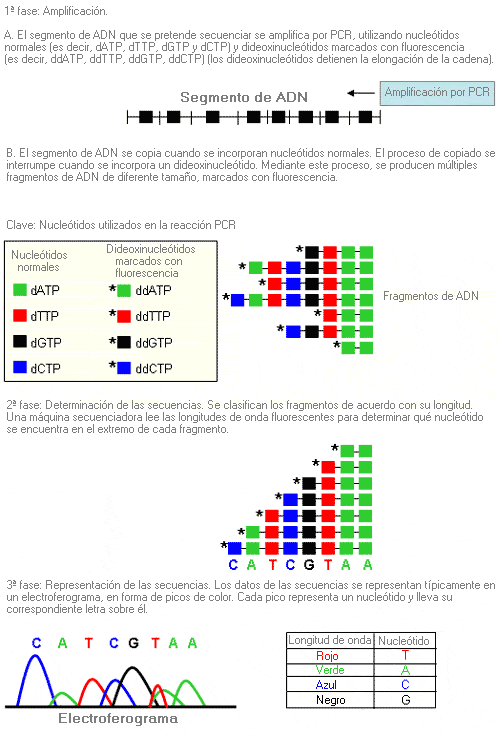
Se puede secuenciar un gen completo, pero la mayoría de las veces se secuencian sólo regiones específicas del gen que muy probablemente contendrán mutaciones (p. ej. los exones y los límites entre los intrones y los exones).
Al secuenciar un segmento de ADN, se identifican la mayoría de las variaciones producidas con respecto a la secuencia normal (por el contrario, el análisis mutacional identifica sólo mutaciones específicas en un segmento determinado de ADN).
Tipos de alteraciones de las secuencias que se pueden detectar:
Alteraciones de secuencia descritas en la literatura como patógenas
Alteraciones de secuencias previsiblemente patógenas, pero no descritas en la literatura
Alteraciones de secuencias de significación clínica imprevisible
Alteraciones de secuencias previsiblemente benignas, pero no descritas en la literatura
Alteraciones de secuencias benignas descritas en la literatura
Posibilidades en caso de que no se detecten alteraciones de las secuencias:
El paciente no presenta una mutación en el gen analizado (p. ej. hay una alteración de la secuencia en otro gen, en un locus diferente)
El paciente presenta una alteración de la secuencia que no se puede detectar mediante el análisis de secuencias (p. ej. deleción de tamaño grande)
El paciente presenta una alteración de la secuencia en una región del gen (p. ej. una región intrónica o reguladora) que no se ha detectado en el análisis de laboratorio
1Adaptado de ACGM Recommendations for Standards for Interpretation of Sequence Variations (2000)
Análisis de truncación de proteínas - Protein truncation testing (sinónimo: PTT)
Método utilizado para identificar las proteínas que se acortan (truncan) a consecuencia de mutaciones que causan específicamente la terminación prematura del proceso de traducción del ARNm.
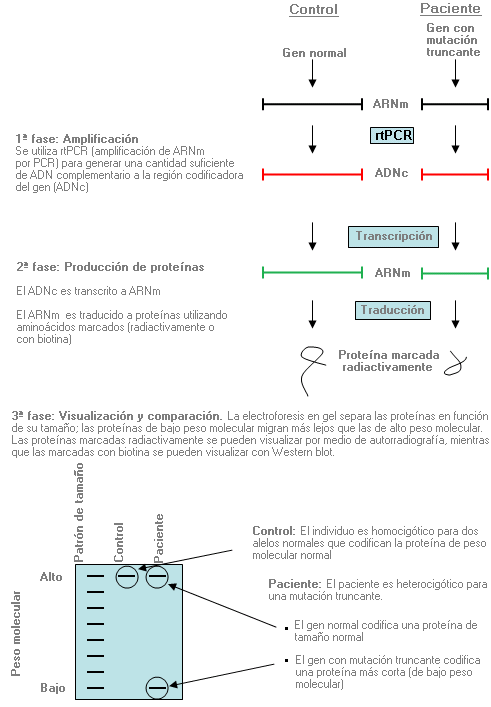
Análisis del haplotipo - Haplotype analysis
Estudio de genética molecular para identificar un conjunto de segmentos de adn que están estrechamente relacionados en su modo de herencia (ligados). Se utiliza en el análisis de ligamiento o cuando un rasgo o característica determinados presentan desequilibrio de ligamiento con un marcador genético o un grupo de [GLOSARIO]marcador[FIN_GLOSARIO]es.
Análisis del origen parental - Parental origen analysis
Análisis directo del ADN - Direct DNA analysis
La utilización de técnicas de secuenciación, análisis mutacional, rastreo de mutaciones o cualquier otro medio de estudio molecular genético, para detectar variantes en un gen que pueden estar asociadas a trastornos genéticos específicos. El análisis directo del adn es posible sólo cuando se conoce el gen o genes o la región genómica, asociados con la enfermedad.
Las técnicas utilizadas para el análisis de ADN directo pueden ser las siguientes:
Análisis de secuencias
Rastreo de mutaciones (p. ej. CSGE, DHPLC, SSCP)
Análisis mutacional (p. ej. PCR, Southern blot, análisis de repeticiones de trinucleótidos, FISH, análisis de truncación de proteínas (PTT), estudios de heterocigosidad, análisis RFLP, ASO)
El análisis de ADN directo contrasta con el análisis de ligamiento (que anteriormente se denominaba análisis de ADN indirecto)
Algunas implicaciones clínicas
El análisis de ADN directo sólo es posible cuando se conoce el gen (genes) o la región genómica asociados con una enfermedad
El análisis de ligamiento se puede realizar cuando no se conoce el gen o los genes asociados con una enfermedad
Análisis mutacional específico - Targeted mutation analysis (sinónimo: análisis mutacional dirigido)
Pruebas para detectar la presencia de una mutación específica (p. ej. Glu6Val en la Anemia de Células Falciformes), un tipo de mutación específica (p. ej. la expansión de trinucleótidos repetidos asociada a la Ataxia Espinocerebelosa tipo 1, deleciones asociadas a la Distrofia Muscular de Duchenne) o un conjunto de mutaciones (p. ej. un grupo de mutaciones en la Fibrosis Quística). Difiere de la secuenciación génica completa o el rastreo de mutaciones, en que éstos detectan la mayoría de las mutaciones en la región investigada.
El análisis mutacional se utiliza en las dos situaciones siguientes:
I. Estudio de enfermedades en las que:
La mayoría o todos los individuos afectados presentan la misma mutación (p. ej. Anemia de Células Falciformes).
La mayoría o todos los individuos afectados presentan el mismo tipo de mutación (p. ej. expansión de repeticiones de trinucleótidos).
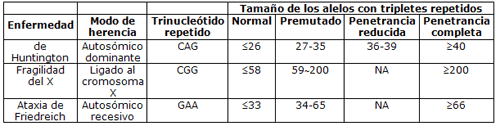
Un grupo de mutaciones son las más comunes y explican un elevado porcentaje de los casos (véase Tabla).

II. Estudio de los miembros a riesgo de una familia tras haber identificado la mutación patológica específica de la familia en un pariente afectado. Por ejemplo, si en el análisis de la secuencia se identifica una mutación patológica en un miembro afectado de la familia, todos los familiares a riesgo deben ser sometidos sólo a un análisis mutacional dirigido a detectar esa mutación específica de la familia.
Algunas implicaciones clínicas
El análisis mutacional no es generalmente el primer estudio de genética molecular que se utiliza para la identificación de mutaciones en enfermedades que se sabe están asociadas con una proporción alta de mutaciones raras (por ejemplo, específicas de una familia).
Si en un individuo afectado no se puede detectar una mutación en el análisis mutacional, puede que sea necesario realizar un análisis de la secuencia o un rastreo de otras mutaciones para identificarla.
Análisis mutacional individualizado - Custom mutation analysis
Estudios de Genética molecular desarrollados especialmente en un laboratorio clínico en función de las necesidades, para detectar una mutación específica que predispone a una enfermedad que no se puede identificar por otros medios clínicos. Las situaciones en las que puede utilizarse un análisis mutacional individualizado son las siguientes: a) estudios de miembros de una familia que tienen riesgo de desarrollar una enfermedad específica, al haberse identificado una mutación patológica en un miembro afectado de esa misma familia; b) estudios prenatales en familias en las que se ha identificado una mutación patológica en un miembro afectado.
Aneuploidía - Aneuploidy
Un aneuploide es un individuo cuyo número de cromosomas difiere del tipo silvestre o euploide en parte de su dotación cromosómica, debido a un cromosoma extra o ausente, que siempre se asocia con una deficiencia en el desarrollo físico, mental o ambos. Generalmente, la dotación cromosómica aneuploide solo difiere de la salvaje en uno o pocos cromosomas. La aneuploidía se puede observar frecuentemente en células cancerosas.
Anotación (relativo a secuenciación) - Annotation
Información adjunta (texto, comentarios, otros) que se añade a una secuencia genómica específica.
Antecedentes familiares - Family history
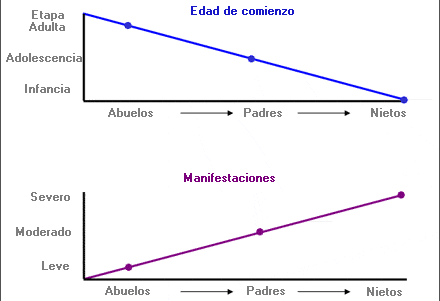
Anticipación - Anticipation
En determinadas enfermedades genéticas, tendencia a que los síntomas de una patología se presenten a edades más tempranas y/o sean más graves que en las generaciones anteriores. Se observa frecuentemente en enfermedades causadas por la expansión de trinucleótidos repetidos que tiende a aumentar de tamaño y a tener un efecto más significativo cuando se transmite de una generación a la siguiente.
Anticodon - Anticodon
Unidad de tres bases de ARN de transferencia, complementaria a un codón en el ARN mensajero.
Anticuerpo - Antibody
Proteína sintetizada por las células plasmáticas en respuesta a un antígeno. Cada anticuerpo se une específicamente a un solo antígeno con el objetivo de destruirlo, bien directamente o bien promoviendo la acción de los glóbulos blancos sobre el mismo.
Antígeno - Antigen
Sustancia ajena al cuerpo que el sistema inmunitario reconoce como “extraña” y que hace que se induzca una respuesta inmunitaria que conlleva la formación de anticuerpos. Los antígenos incluyen toxinas, sustancias químicas, bacterias, virus, etc.
Apoptosis -
Muerte celular programada, la ruta normal por la que el organismo se libera de las células dañadas, no deseadas o innecesarias.
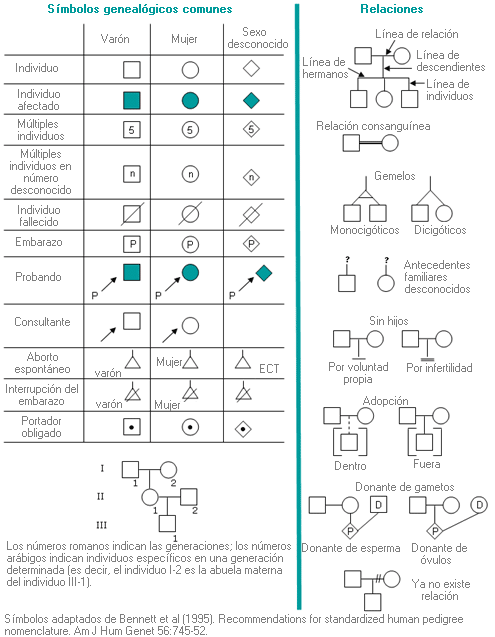
Árbol genealógico - Genealogía - Árbol familiar - Pedigree - Genograma - Pedigree
Arginina - Arginine
La arginina (arg o R) es uno de los 20 aminoácidos que se encuentran formando parte de las proteínas. Es un aminoácido condicionalmente esencial (se necesita en la dieta solo bajo ciertas condiciones), y puede estimular la función inmunológica al aumentar el número de leucocitos. La arginina está involucrada en la síntesis de creatina, poliaminas y en el ADN. En el ARNm está codificado por los codones CGU, CGC, CGA, CGG, AGA, y AGG.
ARN (ácido ribonucleico) - RNA (ribonucleic acid)
El ácido ribonucleico (ARN o RNA) es un ácido nucleico formado por una cadena de ribonucleótidos. Está formado por una cadena simple de ribonucleótidos, cada uno de ellos formado por ribosa, un fosfato y una de las cuatro bases nitrogenadas (adenina, guanina, citosina y uracilo). El ARN celular es lineal y monocatenario (de una sola cadena), pero en el genoma de algunos virus es de doble hebra.
ARN mensajero (ARNm) - Messenger RNA (mRNA)
Es el ácido ribonucleico que transfiere el código genético procedente del núcleo celular a un ribosoma en el citoplasma. Se forma durante el proceso de transcripción del ADN al ARN, durante el cual la ARN polimerasa debe unirse al ADN con ayuda de cofactores de transcripción. El proceso de transcripción se lleva a cabo en el núcleo en eucariotas y en el citoplasma en procariotas.
ARNt - tRNA
ARN de transferencia (ARNt) es una pequeña molécula de ARN que participa en la síntesis de proteínas. Su función es suministrar los aminoácidos al ribosoma para que éste lleve a cabo la síntesis de la proteína según la secuencia especificada de un ARN mensajero. Una vez el ribosoma usa el aminoácido transportado por el ARNt, este se separa del ribosoma y se desplaza por el citoplasma buscando nuevos aminoácidos.
Asesoramiento genético - Genetic counseling
ASO -
Asociación - Association
En epidemiología genética, situación en la que un alelo se encuentra más o menos representado en un grupo de individuos afectos de lo que se esperaría por su frecuencia en la población general.
Asparagina - Asparagine
La asparagina (abreviada como Asn o N) es uno de los 20 aminoácidos codificados en el código genético, y los codones que la codifican son AAU y AAC. En el ser humano no es un aminoácido esencial.
Atlas del Genoma del Cáncer - The Cancer Genome Atlas TCGA
Proyecto internacional iniciado en el año 2005 con el objetivo de catalogar los cambios moleculares, a nivel de ADN, ARN, proteico y epigenético, existentes en los diferentes tipos de tumores mediante plataformas de análisis masivo y potentes análisis bioinformáticos. Con este proyecto se pretende mejorar el diagnóstico y el tratamiento de los pacientes oncológicos además de proporcionar información para prevenir y entender cuáles son los mecanismos moleculares asociados al desarrollo de esta enfermedad (https://cancergenome.nih.gov/)
Autosoma - Autosome
Autosómico - Autosomal
Se refiere a cualquiera de los cromosomas que no son los determinantes del sexo (es decir, X e Y) o a los genes de esos cromosomas.
Autosómico dominante - Autosomal dominant
Término que se utiliza para describir un rasgo o patología asociados a un determinado alelo, que están presentes en todos los individuos que han heredado una sola copia de dicho alelo (heterocigotos). Se refiere específicamente a un gen de uno de los 22 pares de autosomas.
Arbol genealógico que representa el patrón de herencia autosómica dominante.
Probabilidad de que una mutación de un gen autosómico dominante sea transmitida a los descendientes.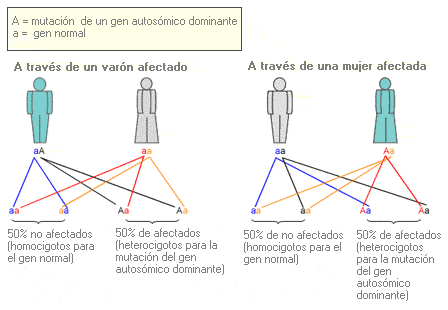
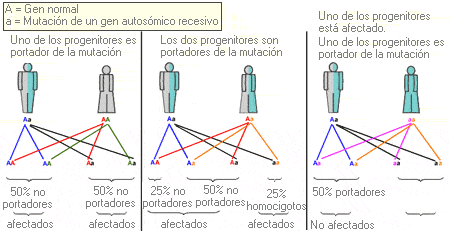
Autosómico recesivo - Autosomal recessive
Término que se utiliza para describir un rasgo o patología que requiere la presencia de las dos copias de un determinado alelo para que se exprese el fenotipo. Se refiere específicamente a los genes de uno de los 22 pares de autosomas.
Árbol genealógico que representa el patrón de herencia autosómica recesiva.
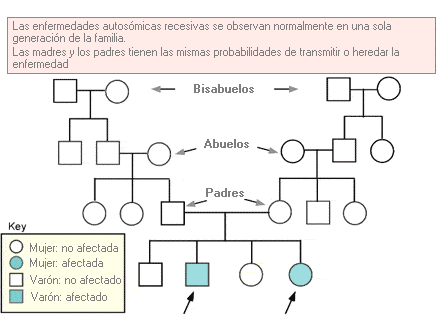
Probabilidad de que una mutación de un gen autosómico recesivo sea transmitida a los descendientes.